癌症是体内异常细胞的不受控制的生长。为了使癌症发生多种信号传导机制,必须分解以允许细胞避免体内的许多检查和平衡,从而防止这种不受控制的生长发生。对支持或防止这种生长的分子相互作用和生物学过程的理解是寻找癌症原因和治愈的关键因素。科学家将分子相互作用和生物过程分为信号传导和调节途径。该信息包括有关途径,构成这些途径的基因以及基因之间相互作用的数据。创建了MDA途径浏览器Web应用程序,以提供一种机制,用户或软件程序可以轻松访问此类信息。它利用PathwaysWeb Web服务从有组织的数据库中检索所需的信息。
最佳MDA途径浏览器是一种仅读取资源的Web应用程序,可以使用标准HTTP协议GET和/或发布请求来检索有关癌症相关的生物途径和相关数据的数据。资源可能是对象(即单个途径)或对象集合(即一组包含某些基因的途径)。可通过应用程序访问的资源包括有关途径,这些途径中的基因,基因之间的相互作用,基因同义词之间的相互作用,对具有批准的Hugo Gene符号的基因的多个基因数据库ID的引用以及与基因和相互作用有关的文章的PubMed ID的引用。该系统包含多个版本的数据,这些数据是从给定时间点的不同来源收集数据的快照。API提供的数据可以通过版本检索以帮助复制结果或轻松地检索最新版本的数据,而无需事先知道最新版本是什么。
MDA途径浏览器Web应用程序资源分类如下:
| 资源类别 | 资源 |
|---|---|
| 基本的 | 途径互动 |
| 元数据 | 途径来源版本互动类型交互向交互PubMed ID列表 |
| 支持数据 | 基因基因名称同义词基因符号同义词Gene PubMed ID列表 |
| 基因本体学相关数据 | 去基因将基因转变为术语到学期关系去分类单元(物种)去期限术语和基因计数去任期和父母 |
最佳途径浏览器资源关系
路径浏览器Web应用程序的功能之一是可以在资源之间导航。pathwaysweb中的最高资源级别是基于从各种来源收集数据的数据版本(资源名称:版本)的集合。版本列表包含版本资源(资源名称:版本)本身包含指向其他资源的链接,例如交互,基因和途径。下图显示了MDA途径浏览器中的资源关系。
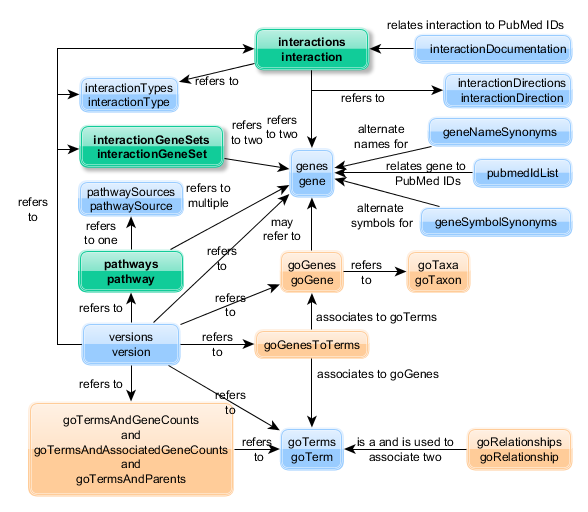 途径浏览器资源关系
途径浏览器资源关系
颜色 资源类型 绿色的 主要资源 浅蓝 元数据和支持资源 杏仁 基因本体相关资源
最佳最佳途径数据
注意:对于给定的途径源,MDA途径ID编号应在数据负载版本之间保持一致。MDA途径ID不一定是顺序数字,通常不等于途径的总数。
途径数据包含以下信息:
MDA路径ID
描述
来自源的ID
源ID
基因数量
基因(HGNC符号)
基因(Entrez ID)
子路径
父途径ID和适当的链接
链接到互动
链接到相关途径图(用于reactome)
版本可选查询参数
查询 /表单参数 参数描述 默认值 开始 起始记录号(第一记录是0) 0 数数 每次呼叫的最大记录数。 20 SourceID 路径源的ID(请参阅pathwaysirces资源以获取可接受的值)。 Allgenes 逗号分隔了雨果基因符号的列表,其中所有基因都必须在路径中。 任何基因 逗号分隔了雨果基因符号的列表,其中至少一个必须在路径中。 阿伦特雷兹 逗号分隔了所有基因都必须在途径中的基因entrez ID列表。 Anyentrezids 逗号分隔了基因entrez ID的列表,其中至少一个必须在路径中。 描述 文本要在描述字段中搜索。(不区分大小写)。 全部 如果应立即发送所有记录,而不是分页,则设置为TRUE。如果存在所有参数,则将忽略启动和计数参数。
途径
返回最新数据库版本中的所有路径名和ID和链接(http get和post)
URL:PathwaysBrowser/Pathways/最新
可选查询/表单参数:开始,count,sourceID,Allgenes,Anygenes,Description,所有
返回指定数据库版本中的所有路径名和ID和链接(http get和post)
URL:pathwaysbrowser/pathways/version/{version_id}
可选查询/表单参数:开始,count,sourceID,Allgenes,Anygenes,Description,所有
路径
返回最新数据库版本中指定ID的途径的所有信息
URL:pathwaysbrowser/pathway/最终/mdapathwayid/{mdapathwayid}
可选查询/表单参数:无
返回指定数据库版本中指定ID的途径的所有信息
URL:pathwaysbrowser/pathway/version/{versionId}/mdapathwayid/{mdapathwayid}
可选查询/表单参数:无
互动
交互数据包含以下信息:
交互ID
交互细节计数
基因a
基因b
Entrez Gene a
Entrez基因b
相互作用方向
类型ID
特定类型
通用类型
是描述性的
积极的
谓词
来源
来源的来源
来自源的文字
PubMed计数
版本可选查询参数
查询 /表单参数 参数描述 默认值 开始 起始记录号(第一记录是0) 0 数数 每次呼叫的最大记录数。 100 基因 逗号分隔的基因符号列表,如果包括列表中的任何基因在左右的互动记录中。不适用于含有途径的URI 限制基因 逗号分隔的基因符号列表必须在此列表中进行交互,以便包括在此列表中。不适用于含有途径的URI 交互式类型 逗号分隔的交互类型ID的列表必须标记为包含在内。(有关有效的交互类型ID,请参见InteractionTypes资源。)不适用于包含pathwayids的URI 全部 如果应立即发送所有记录,而不是分页,则设置为TRUE。如果存在所有参数,则将忽略启动和计数参数。
一阶交互
返回指定数据库版本中的一阶交互信息(http get和post)
URL:pathwaysbrowser/互动/最新
可选查询/表单参数:开始,计数,基因,限制基因,相互作用类型,所有
返回指定数据库中的一阶交互信息(http get和post)
URL:PathwaysBrowser/Interactions/version/{version_id}
可选查询/表单参数:开始,计数,基因,限制基因,相互作用类型,所有返回最新数据库版本中指定途径ID的一阶交互信息
URL:pathwaysbrowser/互动/最新/pathway/pathwayids/{csv pathway ID}}
可选查询参数:开始,计数,所有
返回指定数据库版本中指定途径ID的一阶交互信息(http get和post)
URL:pathwaysbrowser/互动/版本/{version_id}/pathwayids/{csv pathway ids iD}}
可选查询参数:开始,计数,所有
一阶相互作用
返回最新数据库版本中指定交互ID的一阶交互信息
URL:pathwaysbrowser/互动/最新/互动/{Interaction_id}
返回指定数据库版本中指定交互ID的一阶交互信息
url:pathwaysbrowser/互动/version/{version_id}/interactionId/{conteraction_id}
最佳最佳
最佳途径来源
途径源数据包含以下信息:
源ID
描述
版本ID
途径来源
在最新数据库版本中返回所有通路源的信息
URL:pathwaysbrowser/pathwaysources/最新
返回指定数据库版本中途径源的所有信息
URL:pathwaysbrowser/pathwaysources/version/{versionId}
路径源
在最新数据库版本中使用指定源ID返回通路源的所有信息
URL:pathwaysbrowser/pathwaysource/festment/sourceid/{sourceId}
在指定数据库版本中返回使用指定源ID的路径源的所有信息
URL:pathwaysbrowser/pathwaysource/version/{versionId}/sourceId/{sourceId}
最佳
互动类型
交互类型数据包含以下信息:
类型ID
特定类型
通用类型
是描述性的
版本
互动类型
返回最新数据库版本中的所有互动类型
URL:PathwaysBrowser/InteractionTypes/最新
可选查询参数:开始,计数,所有
返回指定数据库版本中的所有互动类型
URL:PathwaysBrowser/InteractionTypes/version/{version_id}
可选查询参数:开始,计数,所有
交互类型
返回最新数据库版本中指定互动类型的信息
URL:pathwaysBrowser/InteractionType/最新/typeId/{Interaction_type_id}
返回指定数据库版本中指定交互类型的信息
URL:pathwaysBrowser/InteractionType/version/{version_id}/typeid/{conteraction_type_id}
最佳最佳基因相关的PubMed ID列表
基因相关的PubMed ID数据包含以下信息:
ID
相关的PubMed ID
版本ID
基因相关的PubMed文档
最新数据库版本中指定的雨果基因ID的PubMed ID列表
URL:pathwaysbrowser/pubMedlist/最新/批准的hugoid/{hugo_gene_id}
指定数据库版本中指定的雨果基因ID的PubMed ID列表
URL:pathwaysbrowser/pubMedlist/version/{version_id}/批准的hugoid/{hugo_gene_id}
最佳相关的PubMed ID列表
相关的PubMed ID数据包含以下信息:
交互ID
互动细节ID
位置(PM = PubMed或PC = PubMed Central)相关PubMed ID
版本ID
相关的PubMed文档
最新数据库版本中指定互动的PubMed ID列表
URL:pathwaysBrowser/InteractionDocumentationList/festment/interactionId/{Interaction_id}
指定数据库版本中指定互动的PubMed ID列表
URL:pathwaysBrowser/InteractionDocumentationList/version/{version_id}/InteractionId/{Interaction_id}
最佳基因
基因数据包括以下信息:
基因符号
基因名称
染色体
Ensembl基因ID
ENSEMBL ID
Entrez基因ID
Entrez基因ID(NCBI)
HGNC ID
基因座组
基因座类型
OMIM ID
refseq
UCSC ID
Uniprot ID
Vega ID
基因家族标签
名称同义词
符号同义词
途径
互动
PubMed ID
版本ID
可选查询参数
查询 /表单参数 参数描述 默认值 开始 起始记录号(第一记录是0) 0 数数 每次呼叫的最大记录数。 100 全部 如果应立即发送所有记录,而不是分页,则设置为TRUE。如果存在所有参数,则将忽略启动和计数参数。
HGNC基因
最新数据库版本中所有基因的Hugo基因符号的返回列表
URL:PathwaysBrowser/Genes/最新
可选查询参数:开始,计数,所有
指定数据库版本中所有基因的雨果基因符号列表
URL:pathwaysbrowser/genes/version/{version_id}
可选查询参数:开始,计数,所有
HGNC基因
最新数据库版本中指定的雨果基因ID的返回基因信息
URL:pathwaysbrowser/gene/最新/批准的hugoid/{hugo_gene_id}
指定数据库版本中指定的雨果基因ID的返回基因信息
URL:pathwaysbrowser/gene/version/{version_id}/批准的hugoid/{hugo_gene_id}
最佳基因名称同义词
基因名称同义数据包含以下信息:
基因符号
名称
版本ID
HGNC基因名称同义词
返回最新数据库版本中指定的Hugo Gene ID的所有各种基因名称
URL:pathwaysbrowser/genEnamesynonyms/最新/批准的hugoid/{hugo_gene_id}
返回最新数据库版本中指定的Hugo Gene ID的所有各种基因名称
URL:pathwaysbrowser/genEnamesynonyms/version/{version_id}/批准的hugoid/{hugo_gene_id}
基因符号同义词
基因符号的同义数据包含以下信息:
基因符号
同义词
版本ID
HGNC基因符号同义词
在最新数据库版本中返回指定的Hugo Gene ID的所有各种基因符号
URL:pathwaysbrowser/genesymbolssynonyms/最新/批准的hugoid/{hugo_gene_id}
返回指定数据库版本中指定的雨果基因ID的所有各种基因符号
URL:pathwaysbrowser/genesymbolssynonyms/version/{version_id}/批准的hugoid/{hugo_gene_id}
返回最新数据库版本中指定基因符号的同义词的所有各种基因符号
URL:pathwaysbrowser/genesymbolssynonyms/最新/同义词/{同义词}
返回指定数据库版本中指定基因符号同义词的所有各种基因符号
URL:pathwaysbrowser/genesymbolssynonyms/version/{version_id}/shonimy/{同义词}
最新数据库版本中指定列表(CSV)的返回批准的基因符号(CSV)
URL:pathwaysBrowser/genesymbolssynonyms/最新/geneids/{CSV基因符号同义词}
指定数据库版本中指定列表指定列表(CSV)的返回批准的基因符号(CSV)
URL:pathwaysbrowser/genesymbolssynonyms/version/{version_id}/geneids/{csv基因符号同义词}
最佳最佳去基因
GO基因数据包含以下信息:
基因符号
基因名称
分类单元
HGNC基因链接
版本ID
可选查询参数
查询 /表单参数 参数描述 默认值 开始 起始记录号(第一记录是0) 0 数数 每次呼叫的最大记录数。 1000 群体 基因的NCBI分类单元(物种)ID。(有效值:9606或10090)。 9606 全部 如果应立即发送所有记录,而不是分页,则设置为TRUE。如果存在所有参数,则将忽略启动和计数参数。 基因本体论基因
检索指定数据库版本中基因本体论中定义的基因信息。
url:/pathwaysbrowser/gogenes/version/{versionId}/
可选查询/表单参数:开始,计数,分类群,所有
检索有关最新数据库版本中基因本体论中定义的基因的信息。
url:/pathwaysbrowser/gogenes/最终/
可选查询/表单参数:开始,计数,分类群,所有
基因本体论基因信息
指定数据库版本中指定分类单元(物种)的最新数据库版本中的基因返回列表(根据GO)。
url:/pathwaysbrowser/gogene/version/{versionId}/genesymbol/{genesymbol}/taxonid/{tualonid}
最新数据库版本中指定分类单元(物种)的指定数据库版本中的基因返回列表(根据GO)。
url:/pathwaysbrowser/gogene/最新/geneSymbol/{genesymbol}/tualonid/{taxonid}
最佳术语和基因
检索与基因本体论中指定术语相关的基因清单。该列表可能包含与HGNC导入的符号的基因符号。如果有相应的HGNC术语,则将显示HGNC的官方HGNC基因符号和基因名称。(有关更多信息,请参见Gogenes资源)。基因本体学相关的基因和术语,但是该GO ID的列表不仅限于GO建立的特定关联。从给定术语回到三个GO本体的根项的所有路径均已确定。该资源返回与特定的GO术语相关的所有唯一基因,无论是通过GO与M d Anderson计算的,使用GO中的信息。
GO期限和基因数据包含以下信息:
去ID
学期
基因
定义
termtype
版本ID
可选查询参数
查询 /表单参数 参数描述 默认值 群体 基因的NCBI分类单元(物种)ID。(有效值:9606或10090)。 9606
基因本体论项和相关基因
检索有关从基因本体论导入的术语以及指定数据库版本中与该术语相关的基因的信息。
url:/pathwaysweb/gotermandgenes/version/{versionId}/goid/{goid}
可选查询/表单参数:分类单元
检索有关从基因本体论导入的术语以及最新数据库版本中与该术语相关的基因的信息。
url:/pathwaysweb/gotermandgenes/最终/goid/{goid}
可选查询/表单参数:分类单元
最佳将基因变为
从基因本体论导入的术语列表与基因本体论中的基因相关。该列表可能包含与HGNC导入的符号的基因符号。(有关更多信息,请参见Gogenes资源)。基因本体学相关的基因和术语,但是该GO ID的列表不仅限于GO建立的特定关联。从给定术语回到三个GO本体的根项的所有路径均已确定。该资源返回与GO关联的每个基因以及沿着词根项相关的所有术语的所有唯一GO ID。
GO基因到术语数据包含以下信息:
基因符号
基因名称
分类单元
HGNC基因链接
相关的GO ID
版本ID
可选查询参数
查询 /表单参数 参数描述 默认值 开始 起始记录号(第一记录是0) 0 数数 每次呼叫的最大记录数。 100 群体 基因的NCBI分类单元(物种)ID。(有效值:9606或10090)。 9606 基因 逗号分开的基因符号列表 全部 如果应立即发送所有记录,而不是分页,则设置为TRUE。如果存在所有参数,则将忽略启动和计数参数。
走上学期协会
检索有关指定数据库版本中指定基因列表的相关基因本体术语的信息。(http获取并发布)
url:/pathwaysbrowser/gogeneStoterms/destry/{destryId}
可选查询/表单参数:启动,计数,分类单元,基因等
检索有关最新数据库版本中指定基因列表的相关基因本体术语的信息。(http获取并发布)
url:/pathwaysbrowser/gogeneStoterms/最新/
可选查询/表单参数:启动,计数,分类单元,基因等
最佳到学期关系
从基因本体论中检索单个术语,该术语用于描述其他术语之间的关系。该术语将与一个基因无关,仅在GO术语之间。
基因本体学术语对术语关系数据包含以下信息:
关系ID
关系术语
版本ID
基因本体学术语到期关系类型
在指定的数据库版本中检索用于将其他GO彼此相关联的GO术语的信息。
url:/pathwaysbrowser/gorelationships/最新/关系/{关系}
在最新数据库版本中检索用于将其他GO彼此相关联的GO术语的信息。
url:/pathwaysbrowser/gorelations/最新基因本体学术语到期关系类型
检索有关在指定数据库版本中相互关联的单个GO术语的信息。
url:/pathwaysbrowser/gorelationship/version/{versionId}/ressentsid/{关系}
检索有关在最新数据库版本中相互关联的单个GO术语的信息。
url:/pathwaysbrowser/gorelationship/最新/关系ind/{ressectionId}
最佳去分类单元
基因本体学分类单元数据包含以下信息:
分类单元ID
通用名称
属
物种
版本ID
基因本体学分类单元(物种)信息
检索从指定数据库版本中从基因本体论导入的分类单元(物种标识符)的信息。
url:/pathwaysbrowser/gotaxa/version/{versionId}/
检索有关最新数据库版本中从基因本体论导入的分类单元(物种标识符)的信息。
url:/pathwaysbrowser/gotaxa/最新/基因本体论单体分类群(物种)信息
检索从指定数据库版本中从基因本体论导入的单个分类单元物种的信息。
url:/pathwaysbrowser/gotaxon/version/{versionId}/taxonid/{tualoNID}
检索有关最新数据库版本中从基因本体学进口的单个分类单元物种的信息。
url:/pathwaysbrowser/gotaxon/最新/taxonid/{tualonid}
最佳去期限
基因本体学术语数据包括以下信息:
去ID
学期
定义
术语类型
版本ID
可选查询参数
查询 /表单参数 参数描述 默认值 开始 起始记录号(第一记录是0) 0 数数 每次呼叫的最大记录数。 20 术语 术语类型的ID。该术语属于基因本体论内特定本体论文本描述的大写字母。
选择是B,C或M,其中B =生物过程,C =细胞成分,M =分子功能全部 如果应立即发送所有记录,而不是分页,则设置为TRUE。如果存在所有参数,则将忽略启动和计数参数。
基因本体论术语
在指定的数据库版本中检索有关多个基因本体术语的信息。
url:/pathwaysbrowser/goterms/version/{versionId}/
可选查询/表单参数:开始,计数,termTypeid,所有
在最新数据库版本中检索有关多个基因本体术语的信息。
url:/pathwaysbrowser/goterms/最新/
可选查询/表单参数:开始,计数,termTypeid,所有基因本体学术语
在指定的数据库版本中检索有关单个基因本体术语的信息。
url:/pathwaysbrowser/goterm/version/{versionId}/goid/{goid}
在指定数据库版本中的最新数据库版本中检索有关单个基因本体术语的信息。
url:/pathwaysbrowser/goterm/最终/goid/{goid}
最佳术语和相关基因
基因本体论项和相关基因数据包括以下信息:
去ID
学期
基因
定义
termtype
版本ID
可选查询参数
查询 /表单参数 参数描述 默认值 群体 基因的NCBI分类单元(物种)ID。(有效值:9606或10090)。 9606 基因本体论项和相关基因
检索有关从基因本体论导入的术语以及与该术语相关的基因以及在指定最新版本中与该术语相关的基因的信息。(http获取并发布)
url:/pathwaysbrowser/gotermsandgenes/version/{versionId}/
可选查询/表单参数:分类单元
检索有关从基因本体论导入的术语以及与这些术语相关的基因数量以及最新数据库版本中与该术语相关的指定基因计数的信息。(http获取并发布)
url:/pathwaysbrowser/gotermsandgenes/最新/
可选查询/表单参数:分类单元
最佳术语和基因计数
基因本体术语和基因计数数据包括以下信息:
去ID
学期
基因计数(合适的并在适当的情况下选择)
定义
termtype
版本ID
可选查询参数
查询 /表单参数 参数描述 默认值 开始 起始记录号(第一记录是0) 0 数数 每次呼叫的最大记录数。 20 群体 基因的NCBI分类单元(物种)ID。(有效值:9606或10090)。 9606 基因 逗号分开基因本体论基因符号。每个GO期限的指定基因的计数将返回。 goids 逗号分隔了所需术语的基因本体学ID列表。
格式:GO:1234567,GO:1234568全部 如果应立即发送所有记录,而不是分页,则设置为TRUE。如果存在所有参数,则将忽略启动和计数参数。 基因本体学术语和所有基因计数
检索有关从基因本体论导入的术语以及与这些术语相关的基因数量以及指定数据库版本中与该术语相关的指定基因计数的信息。(http获取并发布)
url:/pathwaysbrowser/gotermsandgenecounts/version/{versionId}/
可选查询/表单参数:开始,计数,分类单元,基因,goids等
检索有关从基因本体论导入的术语以及与这些术语相关的基因数量以及最新数据库版本中与该术语相关的指定基因计数的信息。(http获取并发布)
url:/pathwaysbrowser/gotermsandgenecounts/astals/
可选查询/表单参数:开始,计数,分类单元,基因,goids等基因本体学术语和相关基因计数
检索有关从基因本体论导入的术语以及与指定数据库版本中这些术语相关的基因总数。(http获取并发布)
url:/pathwaysbrowser/gotermSandAssociatedGeneCounts/destry/{versionId}/
可选查询/表单参数:开始,计数,分类单元,goids等
检索有关从基因本体论导入的术语以及与最新数据库版本中这些术语相关的基因总数。(http获取并发布)
url:/pathwaysbrowser/gotermSandAssociatedGeneCounts/astals/
可选查询/表单参数:开始,计数,分类单元,goids等
执行条款和父条款
基因本体术语和家长术语数据包含以下信息:
去ID
父级ID
关系ID(术语与项关系的ID-每个父术语ID一个)
版本ID
可选查询参数
查询 /表单参数 参数描述 默认值 开始 起始记录号(第一记录是0) 0 数数 每次呼叫的最大记录数。 40000 goids 逗号分隔了所需术语的基因本体学ID列表。
格式:GO:1234567,GO:1234568全部 如果应立即发送所有记录,而不是分页,则设置为TRUE。如果存在所有参数,则将忽略启动和计数参数。 基因本体论术语和相关父母术语
检索有关基因本体术语的信息,相关的父母在指定的数据库版本中。(http获取并发布)
url:/pathwaysbrowser/gotermsandparents/version/{versionId}/
可选查询/表单参数:开始,计数,goids等
在最新数据库版本中检索有关基因本体术语相关的父词的信息。(http获取并发布)
url:/pathwaysbrowser/gotermsandparents/festment/
可选查询/表单参数:开始,计数,goids等
最佳HGNC数据集
使用:用于基因和基因资源,并在路径,路径,相互作用和相互作用资源中提供正确的Hugo基因名称。
描述:http://www.genenames.org/cgi-bin/hgnc_stats.pl
Data: http://www.genenames.org/cgi-bin/hgnc_downloads?col=gd_hgnc_id&col=gd_app_sym&col=gd_app_name&col=gd_status&col=gd_locus_type&col=gd_locus_group&col=gd_prev_sym&col=gd_prev_name&col=gd_aliases&col=gd_name_aliases&col=gd_pub_chrom_map&col=gd_date2app_or_res&col=gd_date_mod&col=gd_date_sym_change&col=gd_date_name_change&col=gd_pub_acc_ids&col=gd_enz_ids&col=gd_pub_eg_id&col=gd_pub_ensembl_id&col=gd_mgd_id&col=gd_other_ids&col=gd_other_ids_list&col=gd_pubmed_ids&col=gd_pub_refseq_ids&col=gd_gene_fam_name&col=gd_gene_fam_pagename&col=gd_record_type&col=gd_primary_ids&col=gd_secondary_ids&col=gd_ccds_ids&col=gd_vega_ids&col=gd_lsdb_links&col=md_eg_id&col=md_mim_id&col=md_refseq_id&col=md_prot_id&col=md_ensembl_id&col=md_ucsc_id&col=md_mgd_id&col=md_rgd_id&status=批准&status =输入+撤回&status_opt = 2&where =&order_by = gd_app_sym_sort&format&format = text&limit =&hgnc_dbtag = on&cistib
ncbi generifs(函数中的基因参考)
使用:用于交互和交互资源。
描述:http://www.ncbi.nlm.nih.gov/projects/generif/generifhelp.html
数据:ftp://ftp.ncbi.nlm.nih.gov/gene/generif/interactions.gz
路径共享:NCI自然SIF(简单交互格式)
使用:用于交互和交互资源。由于条目是蛋白质或其他基因产物而不是基因,因此许多记录被删除。
描述:http://www.pathwaycommons.org/pc/sif_interaction_rules.do
数据:http://www.pathwaycommons.org/archives/pc2/搜索“ pathway commons.7.all.extended_binary_sif.hgnc.sif.gz”
预测网络:CSV
使用:用于交互和交互资源。仅使用功能相互作用的数据源(2010年)和CONSENTION提取(2010年)。从PubMed和Medline开采的相互作用被确定为包含过多的不良数据无法使用。描述:http://www.ncbi.nlm.nih.gov/pmc/articles/pmc3245161/ and https:///compbio.dfci.harvard.edu/predictictioncivenetworks/ data:https:/https://preadiveNiviNetworks/network/download/3?格式= CSV&Extension = CSV和https://compbio.dfci.harvard.harvard.edu/predictivevientetworks/networks/networks/networks/networks/networks/download/4?format = csv&extension = csv&extension = csv = csv = csv = csv = csv = csv = csv = csv = csv = csv
NCBI基因至PubMed
使用:用于基因和基因资源。
描述:http://www.ncbi.nlm.nih.gov/entrez/query/static/static/entrezlinks.html#gene
数据:ftp://ftp.ncbi.nlm.nih.gov/gene/data/gene2pubmed.gz
途径和基因
使用:用于路径,路径和交互资源。
描述:最初从Cabio Web服务进口的那些途径中NCI-NATURE策划和Reactome途径和基因的列表。https://wiki.nci.nih.gov/display/cabio/cabio+Wiki+Home+Page
由于NCI-Nature已停止对此数据的更新,因此该服务现在使用此数据的缓存版本。
数据:使用Cabio Java API从CABIO Web服务中通过客户端软件阅读。
NCI途径和子路径(从网页上刮擦)
使用:用于路径资源。
描述:用于确定CABIO API的路径是否是子路径和父级。
由于NCI-Nature已停止对此数据的更新,因此该服务现在使用此数据的缓存版本。
数据:http://pid.nci.nih.gov/browse_pathways.shtml
Reactome
使用:用于提供Reactome途径,途径描述和途径关系。
描述:途径:http://www.reactome.org/download/current/reactomepathways.gmt.zip
描述:http://www.reactome.org/download/current/reactomepathways.txt
关系:http://www.reactome.org/download/current/reactomepathwaysrelation.txt
基因本体论
使用:用于基因本体相关的Gogenes,Gogene,Gotaxa,Gotaxon,Goterms,Goterms,Goterms,Gorelationships,Gorelationship,GogenEstoterms,GoterMessanterms,GotermSandGeneCounts和GotermSandParents资源。描述:数据的最新sql data的SQL转储从Archive.geneontology.org/latest-lite下载。SQL被过滤以仅检索我们想要的桌子,并且SQL被解析,过滤和重新格式化。
交互搜索术语
使用:在交互和交互资源中使用,以创建转换交互描述为交互类型。
描述:从用于搜索交互详细信息并确定交互类型的多个来源的交互描述中手动创建的列表。
互动类型
使用:用于交互,交互,相互作用类型和交互作用资源。
描述:手动创建的列表用于对交互类型进行分类(某些详细类型和通用类型)。
根据对交互源文件和在线文档中交互描述的审查而创建的。
最佳构建了相互作用类型,以将不同交互信息源提供的各种描述分组为不同交互类型的较小子集。不同来源提供的一些相互作用描述非常描述性,而另一些则不是。没有标准。搜索了各种来源给出的描述,以获取某些关键术语,然后用来编译相当特定的交互类型列表,并将更多的通用交互类型分组。来源提供的一些描述暗示了如何发现相互作用,而不是它是什么类型的相互作用。那些实际描述了相互作用的方式,即可以在is_decriptive字段中识别真正的交互类型,在该字段中分配了y值,在该字段中,当其他人被分配了N的值。分配了交互ID并尝试保留一个尝试它们在数据版本之间保持一致。
交互类型ID 交互类型 互动通用类型 描述性 1 结合 绑定 y 2 binds_to 绑定 y 3 form_bond 绑定 y 4 catalyzes_palmitoylation 催化 y 5 代谢_CATALYSIS 催化 y 6 切割 CLEAVAGE_OF_BOND y 7 脱甲基胺 CLEAVAGE_OF_BOND y 8 去磷酸化 CLEAVAGE_OF_BOND y 9 desumoylates CLEAVAGE_OF_BOND y 10 去泛素化 CLEAVAGE_OF_BOND y 11 state_change 转换 y 12 乙酰化 covalent_modification y 13 Myristoylation covalent_modification y 14 磷酸化 covalent_modification y 15 poly(adp_ribosyl)ates covalent_modification y 16 s_nitrosylation covalent_modification y 17 sumoylation covalent_modification y 18 泛素化 covalent_modification y 19 二聚体 二聚化 y 20 form_diamer 二聚化 y 21 form_homomeric_complex 二聚化 y 22 form_homooligomer 二聚化 y 23 异二聚化 二聚化 y 24 同二聚化 二聚化 y 25 寡聚化 二聚化 y 26 激活 增强 y 27 增强 增强 y 28 表型_enhancement 增强 y 29 properses_ubiquitination 增强 y 30 form_complex form_complex y 31 抑制 抑制 y 32 表型_suplession 抑制 y 33 预防_activation 抑制 y 34 压抑 抑制 y 35 acts_on Reatse_type_unknown y 36 Associates_with Reatse_type_unknown y 37 相互作用 Reatse_type_unknown y 38 互动_WITH Reatse_type_unknown y 39 is_a Reatse_type_unknown y 40 is_covallyse_attached Reatse_type_unknown y 41 neighbouring_reaction Reatse_type_unknown y 42 ubiquitin_ligase_activity covalent_modification y 43 反应 Reatse_type_unknown y 44 reacts_with Reatse_type_unknown y 45 non_interaction 其他 n 46 affinity_capture 其他 n 47 双向 其他 n 48 生化_ACTIVITY 其他 n 49 co_control 其他 n 50 co_crystal_structure 其他 n 51 co_fractionation 其他 n 52 co_localization 其他 n 53 co_purification 其他 n 54 Ecrel 其他 n 55 far_western 其他 n 56 烦恼 其他 n 57 g_erel 其他 n 58 in_same_component 其他 n 59 体外 其他 n 60 in_vivo 其他 n 61 indirect_complex 其他 n 62 PCA 其他 n 63 蛋白_肽 其他 n 64 Protein_RNA 其他 n 65 Recontited_complex 其他 n 66 Synthetic_rescue 其他 n 67 targets_chip 其他 n 68 targets_core 其他 n 69 两_HYBRID 其他 n 70 sequenty_catalysis 其他 n 71 IS_分割 CLEAVAGE_OF_BOND y 72 UBC9与 其他 n 73 no_decription_provided 其他 n 74 同源化 绑定 y 75 functional_interaction 其他 n 76 dosage_rescue 其他 n 77 dosage_lethality 其他 n 78 proximity_label_ms 其他 n 79 synthetic_growth_defect 其他 n
©2013-2018德克萨斯大学医学博士安德森癌症中心